Introducción
Las aves de corral representan una de las principales fuentes de obtención de proteína de origen animal, por esta razón es común encontrar distintas aves domésticas bajo condiciones de traspatio, donde se observan
gallinas (Gallus gallus domesticus) y guajolotes (
Meleagris gallopavo) principalmente, además de patos (Anas platyrhynchos domesticus), gansos (Anser anser) y gallinas de Guinea (Numida meleagris) (Zaldívar-Pedroso, 2007).
En México, las guineas como especie exótica ocupan un área aproximada de 5,648 ha. (SEMARNAT, 2000), siendo reportadas principalmente en unidades de manejo para la conservación de vida silvestre (UMAs) de Coahuila, Hidalgo y Nuevo León (Goméz-de
Silva, et al., 2005).
En la actualidad, la gallina de Guinea ha tomado preferencia en el gusto de las personas para su crianza, ya sea como ave de ornato o productora de carne y/o huevo debido a su rusticidad y el poco manejo. Esta ave tiene su origen en el centro
y este africano, y aunque ya era conocida por griegos y romanos, sólo ha sido utilizada recientemente para la producción de carne (Zaldívar-Pedroso, 2007). Entre las ventajas que ofrece la producción de gallinas de Guinea se encuentra
su rusticidad y buena adaptación a condiciones adversas, cualidades que la convierten en una especie avícola efectiva y novedosa para los productores de traspatio (Gómez-de Silva, et al., 2005; Salgado, et al., 2011), aunque la información
acerca de esta especie en cuanto a la producción es escasa, se conoce que es mantenida en algunas comunidades del país (Camas-Robles, et al., 2017).
Sin embargo, algunas enfermedades originadas por bacterias afectan directamente a las aves, provocando una baja considerable respecto a la producción. La familia Enterobacteriaceae es la más grande y heterogénea de bacilos Gramnegativos
con importancia en la salud humana y animal (Araujo-Guerra, et al., 2019). Dentro de las enfermedades bacterianas más comunes se encuentran
Salmonella spp. y Escherichia coli, las cuales afectan el tracto intestinal del ave, provocando su muerte; adicionalmente, algunos serotipos bacterianos pueden llegar al consumidor a través de la carne o los huevos,
generando un impacto negativo en la seguridad e inocuidad alimentaria (Leotta, et al., 2005; Rincón-Acero, et al., 2011; Araujo-Guerra, et al., 2019).
Respecto a Escherichia coli, muchos serotipos han sido asociados con el síndrome urémico hemolítico en humanos (Ochoa, et al., 2004), siendo el serotipo O157:H7 su principal causante. Por otra parte, las infecciones por Salmonella spp. se relacionan con enfermedades transmitidas por alimentos contaminados (Rincón-Acero, et al., 2011) como la carne, que resultan peligrosos cuando son mantenidos bajo circunstancias que favorecen su multiplicación, situación
que representa un grave problema de salud pública (Bello-Pérez, et al., 1990; Mussaret, et al., 2006).
Si bien la crianza de gallinas de Guinea representa una alternativa eficiente para la producción de alimentos a pequeña escala, es importante realizar estudios para identificar a los microorganismos que ponen en riesgo la salud tanto del animal
así como del consumidor. Por tanto, el objetivo del estudio fue evaluar la prevalencia y caracterizar molecularmente los aislamientos de
Escherichia coli y Salmonella spp. en gallinas de Guinea (Numida meleagris) en dos ranchos de los municipios de Villaflores y La Trinitaria, Chiapas, México.
MATERIALES Y MÉTODOS
El presente estudio se realizó en los ranchos de “Las Amazonas” del municipio de Villaflores, Chiapas, localizado en las coordenadas geográficas 16° 13′ 57″N, 93° 15′ 57″O y “Monte Calvario” del municipio de La Trinitaria, Chiapas, localizado
en las coordenadas geográficas 16° 7′ 13″N, 92° 2′ 27″O. La identificación de bacterias, así como su caracterización molecular, se realizó en el Laboratorio de Biología Molecular de la Facultad de Medicina Veterinaria y Zootecnia (FMVZ)
de la Universidad Autónoma de Chiapas (UNACH).
Se utilizó un muestreo aleatorio sin reemplazo, seleccionando 30 % de la población total de aves en ambos ranchos. Se tomaron muestras por hisopado cloacal de las guineas con apoyo del medio Stuart, posteriormente se colocaron bajo refrigeración
a una temperatura de 4°C hasta su procesamiento. Las muestras se inocularon en cajas de Petri con los medios agar Salmonella Shigella (MCD Lab®) y
Eosiona Azul de Metileno (MCD Lab®) para la identificación de Salmonella spp. y
Escherichia coli respectivamente. Posteriormente se incubaron a 37°C durante 24 h; transcurrido este periodo, se realizó la identificación de las bacterias, siendo positivas para Escherichia
coli cuando las colonias tomaban una coloración verde fosforescente y para Salmonella
spp., cuando las colonias presentaban centros de coloración obscura. Consecutivamente se realizó una resiembra bacteriana, incubando las muestras a una temperatura de 37°C durante 24 h, con la finalidad de aislar las colonias positivas,
posteriormente se trataron con un caldo de Infusión
Cerebro Corazón (MCD Lab®), para evitar la muerte de las colonias dentro del medio agar, posteriormente se homogenizaron con un agitador tipo Vórtex y se incubaron a 37°C durante 24 h, posterior al tiempo transcurrido, se observó
en las muestras una coloración turbia indicando que las colonias crecieron en el caldo; posteriormente, las muestras se inocularon en agar Muller Hinton (MCD Lab®) y se incubaron a 37°C durante 24 h. Consecutivamente, las muestras
se conservaron con glicerol y se llevaron a una temperatura de -26°C.
Posteriormente, se realizó el proceso de extracción de ADN siguiendo la metodología utilizada por Leotta, et al. (2005), donde en agua destilada estéril se colocó una pequeña colonia bacteriana, las muestras se homogenizaron y se trasladaron
a una incubadora de baño seco (Thermo Shaker® TSI-100, USA) a una temperatura de 100°C durante 15 min. con 600 rpm; posteriormente, las muestras se centrifugaron con una centrífuga (VelaQuin® VE-4000, México) a 1500 rpm durante 5 min.,
posteriormente, se extrajo el sobrenadante y se analizó la cantidad de ADN en un espectrofotómetro nanodrop (Thermo Scientific®, ND-ONE-W, USA).
Para la identificación del gen rfbO157 de Escherichia coli, se utilizó la técnica de reacción en cadena de la polimerasa (PCR punto final), colocando en tubos para microcentrífuga, 6.5 μL de agua estéril, 2.5 μL del primer forward,
2.5 μL del primer reverse (utilizando las secuencias de nucleótidos descrita por Leotta, et al., 2005 que se muestran en la tabla 1), 12.5 μL de solución premezclada con Taq ADN polimerasa, dNTP, MgCl2 y tampones de reacción (GoTaq® Master
Mix) y 1 μL de ADN por muestra; las muestras se colocaron en un termociclador (Bio Rad® C-1000, USA) bajo los siguientes ciclos: 94°C durante 5 min, 30 ciclos a 94°C durante 30 s, 52°C durante 30 s, 72°C durante 1 min, con un volumen total
de 25 µL a 105°C.

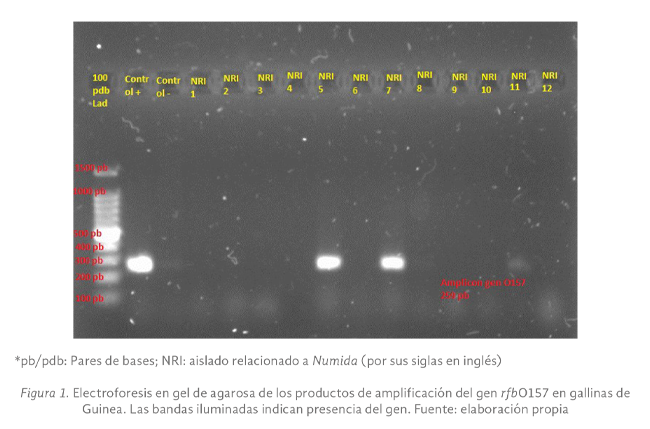
La detección de las bandas fue mediante gel de agarosa al 2% teñido con bromuro de etidio, colocándose en cada pozo 1 μL de azul de bromofenol y 4 μL de ADN, además de colocar en el primer y segundo pozo la referencia positiva y negativa del
gen respectivamente; la cámara de electroforesis se programó a 60 V durante 45 min. La visualización de los resultados de electroforesis (Figura N° 1) se realizó con luz UV en un transiluminador (Bio Rad® Chemi-Doc XRS+, USA).
Los datos se analizaron mediante fórmula de prevalencia puntual (Rodríguez-Hernández, 2015), como se muestra a continuación:
Prevalencia puntual= Ct / Nt
Donde:
Ct: Número de casos existentes (prevalente) en un momento o edad determinados.
Nt: Número total de individuos en la población en ese momento o edad determinados.
RESULTADOS Y DISCUSIÓN
No se encontró prevalencia de Salmonella spp. en ninguno de los ranchos estudiados, resultados similares a los de García, et al. (2009), quienes realizaron un estudio en España con gallinas ponedoras, encontrando una prevalencia
de 1.3% de Salmonella spp. en heces colectadas mediante hisopado cloacal; esto demuestra que la prevalencia de Salmonella es baja en este tipo de método de muestreo; sin embargo, estos resultados difieren a los obtenidos
por Castañeda-Salazar, et al. (2018), quienes encontraron en pollos destinados para consumo humano en Colombia una prevalencia de 29.2%
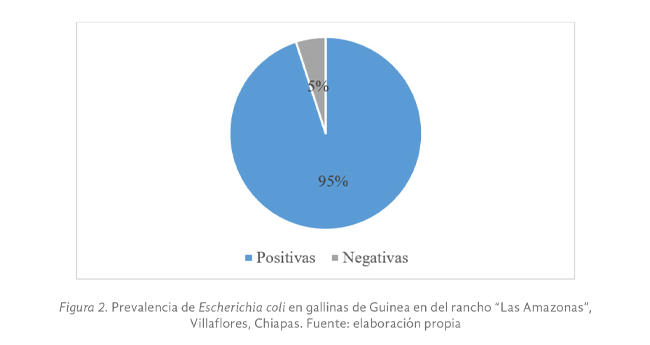
En el caso de Escherichia coli, no se encontró prevalencia en el rancho “Monte Calvario” de La Trinitaria, mientras que en el rancho “Las Amazonas” de Villaflores, el 95% de las muestras fueron positivas (Figura N° 2), esto se atribuye
al tipo de manejo empleado con las guineas y a la variación de temperaturas entre los municipios, hallazgos de Hernández-Fillor, et al. (2017), quienes realizaron un estudio en Cuba con gallinas ponedoras y encontraron una prevalencia
del 45.9% que difieren con este estudio; por otra parte, Gibert-Perelló, en 2010, encontró una prevalencia de 72.7% en muestras de distintos órganos provenientes de gallinas ponedoras en España. Sin embargo, para la identificación del
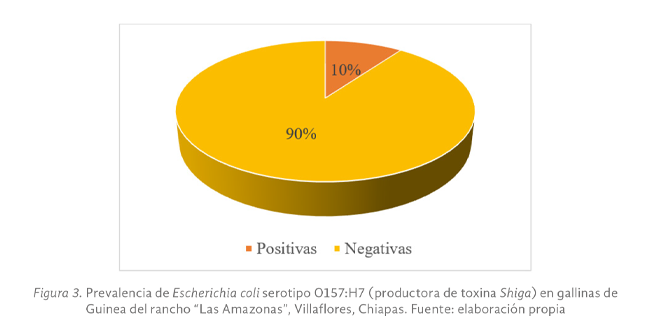
serotipo O157:H7 (productor de toxina Shiga), se encontró el 10% de prevalencia (Figura N° 3), esto difiere con los hallazgos de Zotta, et al. (2016), quienes no reportaron presencia del gen en vísceras de pollos en Argentina; un
estudio realizado por Rípodas-Navarro, et al. (2017), encontró el 12.12% de presencia del gen en carnes y sus derivados de especies domésticas de abasto en Madrid, España.
Estudios han demostrado la capacidad de Escherichia coli O157:H7 de colonizar la mucosa intestinal de gallinas y diseminarse al ambiente (Best, et al., 2005; La Ragione, et al., 2005), lo cual puede explicar que las gallinas y las guineas
pueden actuar como portadoras de Escherichia
coli O157:H7, siendo una posible amenaza a la salud pública.
CONCLUSIÓN
La crianza de gallinas de Guinea representa una alternativa para productores de traspatio debido a la fácil adaptación al medio, así como la poca inversión inicial, además de presentar una fuente para la obtención de proteína de origen animal.
Si bien no se encontró prevalencia de Salmonella spp. en guineas, el estudio demuestra que las gallinas de guinea son portadoras de Escherichia coli O157:H7, por lo que es preciso seguir con estudios del impacto del serotipo
en la sanidad animal, así como en la salud pública.
REFERENCIAS
Araujo-Guerra, A., Fragozzo-Castilla, P., Orlando-Pineda, Á., Mejía-Arbeláez, J. y Peña-Güillen, A. (2019). Detección de Enterobacteriaceae y Hongos en huevos frescos de gallina para el consumo
humano en la Ciudad de Valledupar, Cesar. Colombia. Vet. Arg. 36(373). ISSN 1852-317X.
Bello-Pérez, L. A., Ortiz-Dillanes, D. M., Pérez-Memije, E., Castro-Domínguez, V. (1990). Salmonella en carnes crudas: Un estudio en localidades del estado de Guerrero. Salud pública Mex. 32,74-79.
Best, A., La Ragione, R., Sayers, A. y Woodward, M. (2005). Role for flagella but not intimin in the persistent infection of the gastointestinal tissues of specific pathogen free chicks by Shiga toxin negative Escherichia coli O157:H7.
Infection and immunity, 73:1836-1846.
Camas-Robles, G.G., Ruíz-Sesma, B., Cigarroa-Vázquez, F.A. y Mendoza-Nazar, P. (2017). Caracterización de la producción intensiva de gallina guinea (Numida meleagris). Revista Mexicana
de Agroecosistemas, 4(2), 104-108.
Castañeda-Salazar, R., Pereira-Bazurdo, A.N., Pulido-Villamarín, A.P., Mendoza-Gómez, M.F. (2018). Estimación de la prevalencia de Salmonella spp. en pechugas de pollo para consumo humano provenientes de cuatro localidades de Bogotá-Colombia.
Infectio 23(1), 27-32.
García, C., Catalá-Gregorio, P., Soriano, J.M., Tudón, A.L., Benítez, V., Andreu, L. y Granero, I. (2009). Salmonella spp. en hisopos cloacales, heces y huevos de gallinas ponedoras: Estudio
preliminar. XLVI Symposium científico de avicultura,191-199.
Gibert-Perelló, M. (2010). Detección y caracterización de aislados de Escherichia coli de origen
clínico y fecal en gallinas ponedoras. (Tesis doctoral, Universidad Complutense de Madrid).
https://www.visavet.es/data/tesis/deteccion-y-caracterizacion-de-aislados-de%20Escherichia-coli-de-origen-clinico-y-fecal-en%20gallinas-ponedoras.pdf
Gómez-de Silva, H., Oliveras-de Ita, A. y Medellín-Legorreta, R.A. (2005). Numida meleagris.
Vertebrados superiores exóticos en México: diversidad, distribución y efectos potenciales.
Instituto de Ecología, Universidad Nacional Autónoma de México. Bases de datos SNIB-CONABIO. Proyecto U020. México. D.F.
Hernández-Fillor, R.E., Báez-Arias, M., Alfonso-Zamora, P. y Espinosa-Castaño, I. (2017). Susceptibilidad antimicrobiana y formación de biopelícula en aislados de Escherichia coli procedentes de gallinas ponedoras. Rev. Salud Anim,
39(3),1-13.
La Ragione, R., Best, A., Sprigings, K., Liebana, E., Woodward, G., Sayers, A. y Woodward, M.J. (2005). Variable and strain dependent colonosation of chickens by Escherichia coli O157.
Veterinary microbiology, 107:103-113.
Leotta, I., Chinen, S., Epszteyn, E., Miliwebsky, I., Melamed, M., Motter, M. (2005). Validación de una técnica de PCR múltiple para la detección de Escherichia coli productor de toxina shiga. Rev
Argent Microbiol 37(1), 1-10.
Mussaret, Z., López-Macías, C., Calva, E. (2006). Estudios mexicanos sobre Salmonella: epidemiología, vacunas y biología molecular. Revista Latinoamericana de microbiología, 48(2), 121-125.
Ochoa, T.J., Salazar-Lindo, E., Cleary, T.G. (2004). Evaluation of children with persistent infectious diarrhea. Semin Pediatr Infect Dis, 15(4), 229-36.
Rincón-Acero, D.P., Ramírez-Rueda, R.Y. y Vargas-Medina, J.C. (2011). Transmisión de Salmonella entérica a través de huevos de gallina y su importancia en salud pública. Salud UIS, 43(2), 167-177.
Rípodas-Navarro, A., Fernández-Moreira, D. y Macho-Martínez, M. (2017). Investigación de Escherichia coli productor de toxinas Shiga (STEC) en carnes y derivados cárnicos. Sanidad mil, 73(3), 147-152
Rodríguez-Hernández, R. (2015). Prevalencia y caracterización molecular de Salmonella spp, en
granjas avícolas de postura comercial en el departamento del Tolima. (Tesis de Maestría, Universidad de Tolima).
http://repository.ut.edu.co/bitstream/001/1690/1/PREVALENCIA%20Y%20CARACTERIZACI%c3%93N%20MOLECULAR%20DE%20SALMONELLA%20SPP%2c%20EN%20GRANJAS%20AV%c3%8dCOLAS%20DE%20POSTURA%20COMERCI.pdf
Salgado, E.R., Rojas, F.P., Martínez, N., Fernández, M.S. y de la Vega, D.M. (2011). Producción extensiva de" pintadas" o gallinas de Guinea en Extremadura. Selecciones Avícolas, 53(3), 61-64.
Secretaría de Medio Ambiente y Recursos Naturales. (2000). Base de datos electrónica del Sistema
de Unidades de Manejo, Conservación y Aprovechamiento de la Vida Silvestre UMA. Reporte interno de la Dirección General de Vida Silvestre, SEMARNAT. México, D.F.
Zaldívar-Pedroso, I.J. (2007). Manual de avicultura. II Edición. SOCPA.
Zotta, C.M., Lavayén, S., Nario, F., Piquín, A. (2016). Detección de Escherichia coli en vísceras de animales bovinos y pollos destinadas para el consumo humano. J. Selva Andina Res. Soc., 7(1), 2-9.

 https://orcid.org/
https://orcid.org/




